확률
- 독립사건: 사건 A가 발생하고 그 다음 사건 B가 발생할 때 선행 사건의 결과가 후행 사건의 결과에 영향을 미치지 않는 경우
- 종속사건: 선행 실험의 결과가 후행 실험의 결과에 영향을 미치는 경우
- 조건부확률 : 사건 A가 발생한 상태에서 사건 B가 발생활 확률 = P(B|A)
- P(B|A) = P(A∩B) / P(A) 이다. (단, 독립사건의 경우 P(B|A) = p(B) 이다.)
- 베이즈 이론: 추론대상의 사전확률과 추가적인 정보를 기반으로 해당 대상의 사후 확률을 추론하는 통계적 방법
(경험을 바탕으로 추론한다는 점이 머신러닝/ 딥러닝과의 공통점이어서 많이 사용된다.)
- P(B|A) = P(A|B)*P(B) / P(A) 이다.
- P(A∩B) = P(A)P(B|A) 에서 사건 B에 관한 식인 P(A∩B) = P(B)P(A|B)로 나타낼 수 있다.
- 따라서 P(A)P(B|A) = P(B)P(A|B) 이므로 베이즈 정리식을 유도할 수 있다.
기본적인 확률에서의 베이즈 이론을 기반으로 사용되는 것이 나이브 베이즈 분류이다.
나이브 베이즈 분류 Naive Bayes Classification
확률분류기의 일종으로 특성들 사이에 독립을 가정한다.
일반적으로 조건적 사건 A에 있어서 세부적인 특성이 존재할 수 있으며 이들의 특성은 서로 균등하고 독립적이라고 가정한다.
P(A1,A2,...An) = P(A1) * P(A2) * .... * P(An)
종류
- 가우시안 나이브 베이즈 : 설명 변수가 연속형인 경우
- 멀티노미얼 나이브 베이즈: 설명 변수가 범주형인 경우
- 베르누이 나이브 베이즈 : 설명변수가 (범주형이면서) 이분형인 경우
[실습] 붓꽃 분류
텍스트 데이터 처럼 희소한 고차원인 경우 높은 정확도와 속도를 제공한다.
(차원이 많다 = 특성값이 많다를 의미 / 희소하다 = 컬럼의 값이 대부분 비워져 있는 것)
적용 분야: 스팸 메일 분류, 문서(주제) 분류, 컴퓨터 네트워크 침입자 분류
패키지 로딩
from sklearn.datasets import load_iris
from sklearn.naive_bayes import GaussianNB
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score, confusion_matrix가우시안 나이브 베이즈 알고리즘(설명 변수가 연속형)을 사용한다.
데이터 로딩 및 분할
x,y = load_iris(return_X_y = True)
x_train, x_test, y_train, y_test = train_test_split(x,y,test_size= 0.3,
random_state= 10, stratify= y)sikitlearn 에 들어있는 데이터는 'return_X_y = True' 를 통해 독립변수와 종속변수의 분할이 가능하다.
(단, 독립변수는 대문자를 이용)
모델 생성 및 평가
혼동행렬을 통해 완벽하게 분류된 데이터임을 알 수 있다.
model = GaussianNB()
model.fit(x_train,y_train)
y_hat = model.predict(x_test)
con_mat = confusion_matrix(y_test,y_hat)
print(con_mat)[[15 0 0]
[ 0 15 0]
[ 0 0 15]]
[실습] 독버섯 분류하기
설명변수가 모두 범주형 자료이다.
= 가우시안 나이브 베이즈 알고리즘이 아닌 멀티노미얼 나이브 베이즈 알고리즘을 사용한다.
패키지 로드
from sklearn.naive_bayes import MultinomialNB
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score, confusion_matrix,roc_auc_score,precision_score
데이터 로딩및 확인
import pandas as pd
import numpy as np
df = pd.read_csv('./dataset/mushrooms.csv')
print(df.shape)
display(df.head())
df.info()(8124, 23)
데이터 인코딩 및 분리
데이터는 범주형인 설명변수는 두 가지 방법으로 인코딩이 가능하다.
1. 원핫 인코딩 2. 라벨인코딩
데이터 분할 먼저 처리한다.
x = df.drop('type', axis= 1)
y = df['type']
1. 원핫 인코딩 방법 (pandas 의 one-hot encoder)
판다스를 사용하면 get_dummies() 로 빠르고 간편하게 인코딩이 가능하다.
print(x.shape, y.shape)
x = pd.get_dummies(x)
print(x.shape)
x.head() # 본인이 해당되는 값만 True 나머지는 False(8124, 22) (8124,)
(8124, 117)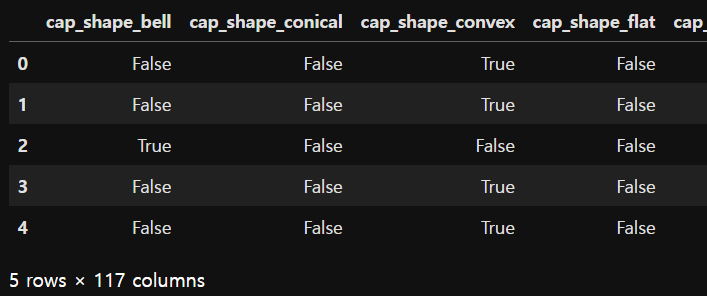
희소행렬로 반환하기 때문에 칼럼의 개수가 늘어난 것을 알 수 있다.
(자신만 True, 나머지는 False 로 반환하며, 자동적으로 컬럼명을 지정해준다.)
y = y.map({'edible': 0, 'poisonous':1})
y.head()0 1
1 0
2 0
3 1
4 0맵핑을 통해 edible = 0, poisonous = 1 로 표현되었다.
훈련, 평가 데이터 분리
x_train, x_test, y_train, y_test = train_test_split(x,y,test_size=0.2,random_state= 10 , stratify= y)
# 분류모형이기 때문에
# 결과값과 비교 위해
print(x_train.shape, x_test.shape)
print(y_train.shape, y_test.shape)
(1) 모델 생성 및 모델 평가
model = MultinomialNB()
model.fit(x_train,y_train)
model.classes_ # 분류할 종류를 보여줌array([0, 1], dtype=int64)
실제값과 예측값 비교할 수 있다.
y_hat = model.predict(x_test)
print('실제값:', np.array(y[:5])) # 바꾸지 않아(인코딩하지 않아) 문자 형태로 나오는 것
print('예측값:', y_hat[:5])실제값: [1 0 0 1 0]
예측값: [0 0 1 0 0]
분류 정확도가 높은 것을 알 수 있다.
cf_mat = confusion_matrix(y_test,y_hat)
print(cf_mat)
print(f'정확도: {accuracy_score(y_test,y_hat):.3f}')
print(f'정밀도: {precision_score(y_test,y_hat):.3f}')
print(f'AUC: {roc_auc_score(y_test,model.predict_proba(x_test)[:,1]):.3f}')[[841 1]
[ 74 709]]
정확도: 0.954
정밀도: 0.999
AUC: 0.997
2. 라벨 인코딩 방법 (scikit-learn 의 LabelEncoder)
apply 함수를 이용하여 익명함수 lamda를 활용하여 컬럼별 라벨 인코딩을 수행하였다.
from sklearn.preprocessing import LabelEncoder
x = x.apply(lambda col: LabelEncoder().fit_transform(col))
print(x.head())cap_shape cap_surface cap_color bruises odor gill_attachment \
0 2 3 0 1 7 1
1 2 3 9 1 0 1
2 0 3 8 1 1 1
3 2 2 8 1 7 1
4 2 3 3 0 6 1
...
[5 rows x 22 columns]
- pandas map을 이용한 라벨인코딩
def labeling(col) : # 컬럼값을 넣음
map_data = { v:i for i,v in enumerate(np.sort(col.unique()))} # 컬럼값의 고유값을 알파벳순으로 정렬
return map_data
x = x.apply(lambda col: col.map(labeling(col)))매핑데이터를 만드는 labeling 함수를 만들어 컬럼값의 고유값을 알파벳순으로 정렬한 후 컬럼의 고유값을 인덱스와 함께 넘겨주었다. 이후 apply 를 이용하여 lambda 함수를 사용하는 것은 위의 방식과 유사하다.(함수를 썼다는게 차이점?)
x_train, x_test, y_train, y_test = train_test_split(x,y,test_size=0.2,random_state= 10 , stratify= y)
# 분류모형이기 때문에
# 결과값과 비교 위해
print(x_train.shape, x_test.shape)
print(y_train.shape, y_test.shape)(6499, 22) (1625, 22)
(6499,) (1625,)원핫 인코딩과 반환되는 열값이 다른 것을 알 수 있다.
(2) 모델 생성 및 평가
label을 따로 매핑하지 않았기 때문에 문자형태로 분류한다.
model = MultinomialNB()
model.fit(x_train,y_train)
y_hat = model.predict(x_test)
print('실제값:', np.array(y[:5])) # 바꾸지 않아(인코딩하지 않아) 문자 형태로 나오는 것
print('예측값:', y_hat[:5])실제값: ['poisonous' 'edible' 'edible' 'poisonous' 'edible']
예측값: ['edible' 'poisonous' 'poisonous' 'edible' 'edible']
labels= ['poisonous','edible'] 를 설정하여 독버섯의 값을 예측한 값부터 나오게 설정
cf_mat = confusion_matrix(y_test,y_hat, labels= ['poisonous','edible']) # labels에 따라 값이 다르게 나옴
print(cf_mat)
print(f'정확도: {accuracy_score(y_test,y_hat):.3f}')
print(f'정밀도: {precision_score(y_test,y_hat, pos_label = "edible"):.3f}') # pos_label 을 설정해주어야함
print(f'AUC: {roc_auc_score(y_test,model.predict_proba(x_test)[:,1]):.3f}')[[611 172]
[ 95 747]]
정확도: 0.836
정밀도: 0.813
AUC: 0.902라벨인코딩
정확도: 0.836 / 정밀도: 0.813 / AUC: 0.902
원핫 인코딩
정확도: 0.954 / 정밀도: 0.999 / AUC: 0.997
= 해당 독버섯 관련 데이터셋에서는 원핫 인코딩 방식으로 데이터를 전처리 하는 것이 정확한 결과값을 얻을 수 있다는 것을 알 수 있었다.
'풀스택 개발 학습 과정 > 머신러닝, 딥러닝' 카테고리의 다른 글
| [머신러닝] 분류분석 : 의사 결정 트리, 앙상블 러닝 - Bagging (0) | 2023.11.22 |
|---|---|
| [머신러닝] 분류분석 : K-NN (0) | 2023.11.22 |
| [머신러닝] 분류분석 : [다지분류]로지스틱 회귀분석 (0) | 2023.11.21 |
| [머신러닝] 분류분석 : [이지분류] 로지스틱 회귀분석 - (실습) 유방암 확률 예측, 개인 신용도 기반 대출 가능 여부 예측 (0) | 2023.11.21 |




